DNA NanotechnologyCOMPANY
DNA ナノテクノロジー
DNAナノテクノロジーとは、DNAの配列特異的な分子集合と周期的な構造を使い、ナノスケールの構造体を設計・作成する技術を基盤とした革新的な分子テクノロジーです。この技術では構造体の作成の柔軟性に加え、機能化が容易であるため、極小空間での分子の振る舞いを観測するナノサイエンスといった基礎分野から、先端計測、材料科学、生命科学、医療、環境エネルギーなど応用分野まで多方面で研究が展開しています。このDNAナノテクノロジーは、分子の自己集合のみを利用してナノスケールの構造体を自在にデザインし作成できる革新的な技術です。ここ20年の間に欧米を中心に急速に発展している新興の学際分野で、コンピューターサイエンス、材料化学、応用物理学、細胞生物学、生物物理学、工学とさまざまな分野の研究者が参入しています。(残念ながら日本では認知度はいまひとつですが)また、研究成果はNature, Scienceとその姉妹紙など世界のトップ科学誌に数多く掲載されています。
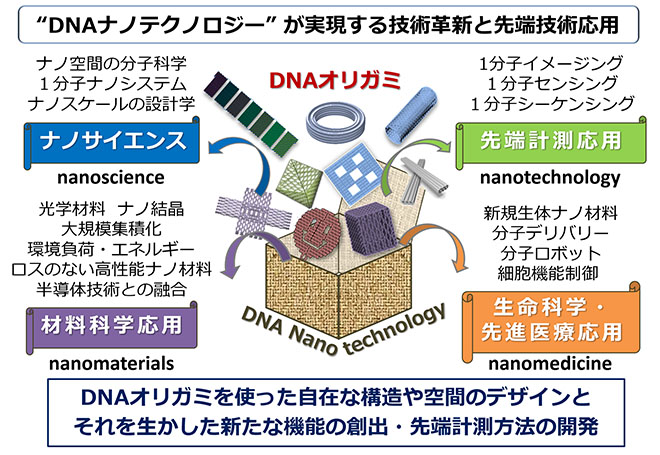
DNAナノテクノロジーの進展によって実現される技術革新
エンジニアリングの分野でもトップダウン型の機械工学や電子工学は製造装置など加工技術が必要であり、また化学分野でも分子の多工程の合成技術が必要です。一方で、生物や細胞は分子の自己集合をもとにあらかじめ決められた集合体を形成し機能を獲得できます。この自己集合をもとにしたものがボトムアップ型の技術です。中でもDNAは超分子として、塩基対形成や配列特異的な2本鎖形成、明確な二重らせん構造をもち、4つの塩基からなる配列のプログラム性、正確な相補鎖形成や予測できる構造、合成のしやすさなどのメリットからボトムアップ型の技術の筆頭であり、「DNAナノテクノロジー」として大きく発展してきました。
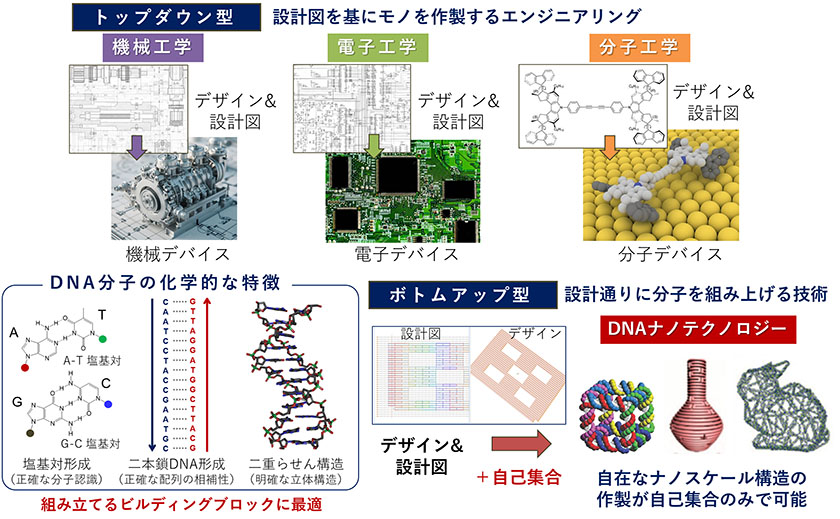
トップダウン型のテクノロジーとボトムアップ型のテクノロジー
DNAナノテクノロジーとは?
DNAナノテクノロジーの源流は、1982年にニューヨーク大学のSeemanが4方向に分岐するDNA構造(Holiday Junction)を自己集合させることで格子構造を作成する方法を考案するところから始まりました。この方法では、4方向に延びる2本鎖DNAの末端に相補的な1本鎖DNA(Sticky end)を使い、配列特異的に末端同士を結合させ、自己集合させることで規則的な2次元の格子構造を作成するというのものです。その後、DNAの自己集合によってキューブ構造などの3次元構造も作成されました。1998年、2本の2本鎖DNAを2箇所で結合させた構造体(Double crossover)を用いて作成したタイル状のDNA構造体を自己集合させることで、原子間力顕微鏡(AFM)でも観察できるサイズの2次元集合体の作成に成功しました。このブレークスルーにより、DNAの配列特異的な自己集合から構造体を作り上げる「DNAナノテクノロジー」の分野が誕生しました。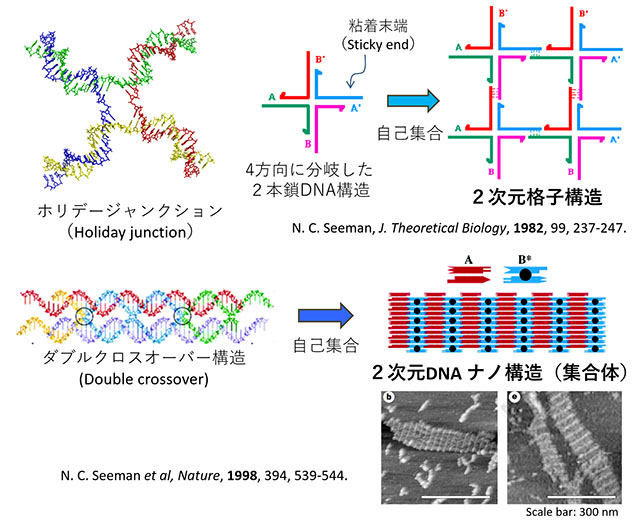
DNAオリガミとは?
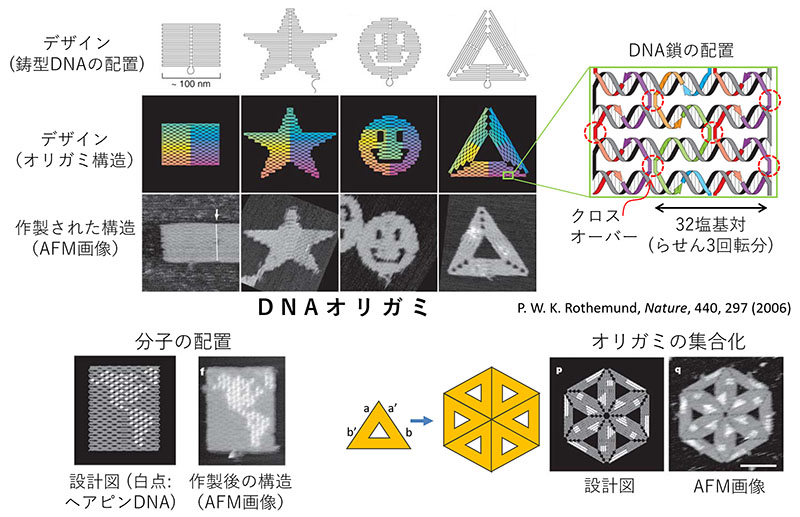
DNAオリガミとは、DNAの自己集合によって形成されるナノスケールの2次元構造体のことです。2006年にカリフォルニア工科大学のRothemundによって開発されました。100
nm程度のナノスケール構造体を自在に設計でき、その作製を行うことができます。DNAオリガミは、2本鎖DNAがクロスオーバーと呼ばれるつながり部分を介して、規則的に架橋して、複数の2本鎖DNAが隣接して配置されることで2次元構造を形成します。また、DNAオリガミはその場所によって異なる配列のDNA鎖が埋め込まれているため、任意の分子を自由な位置に配置することも可能です。構造の外周にステープル鎖を介してDNAオリガミを集合化することも可能です。
その後、2本鎖DNA同士のクロスオーバーを平面方向に対して垂直方向を加えることで3次元のDNAオリガミ構造体の構築方法も確立されました。また、2本鎖DNAが6本配置されたハニカム構造を基本とする3次元構造もデザインでき、その作製も行うことができます。こちらもクロスオーバーを240度の角度を持って架橋することで安定な3次元構造を実現しています。
DNAオリガミ
3次元DNAオリガミ
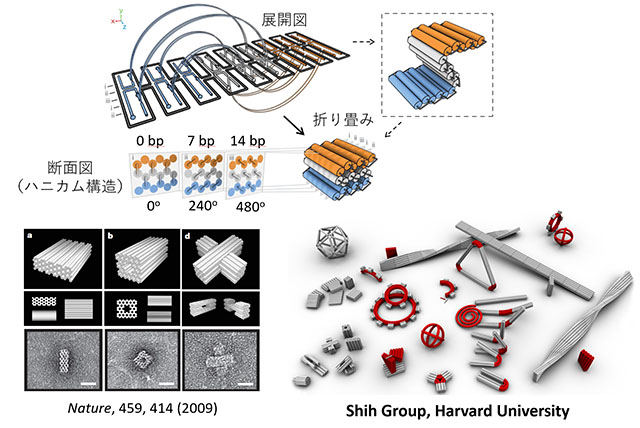
DNAオリガミの設計と作成方法
DNAオリガミは思い通りのナノスケール構造をデザインでき、作製できる手法です。そのためにまずは構造の設計図が必要です。作製したい構造の形状とサイズをデザインし、DNAオリガミ設計用のソフトウエアcaDNAnoを用いて具体的な設計図を作成します。鋳型になるDNAの塩基数に制約は受けますが、鋳型DNA鎖を一筆書きに配置し、その相補鎖を付け、ステープルDNAを規則に従って切ります。完成したDNAオリガミのステープルDNAの配列は配列表として取り出せます。ステープルDNA(多くは32量体,220本程度)はDNA製造会社に作製してもらい、それらすべてを混合します。鋳型となる環状の1本鎖DNA(多くはM13mp18環状一本鎖DNA,7249塩基)に混合したステープルDNAを加え、加熱し、ゆっくり冷却(アニーリング)し自己集合させます。これにより、あらかじめ設計したDNAナノ構造体と同じ形状・サイズのDNAオリガミを高収率で作製することができます。構造の確認は平面の場合は原子間力顕微鏡(AFM)、立体の場合は電子顕微鏡が用いられます。
1. DNAオリガミのデザインと設計法
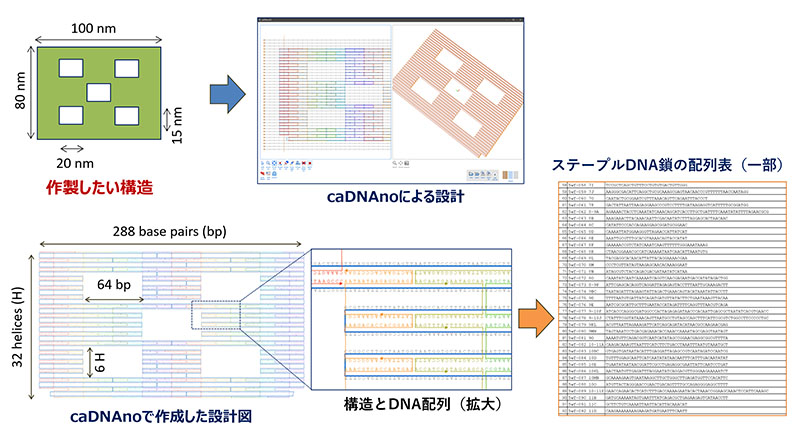
2. DNAオリガミの作成方法
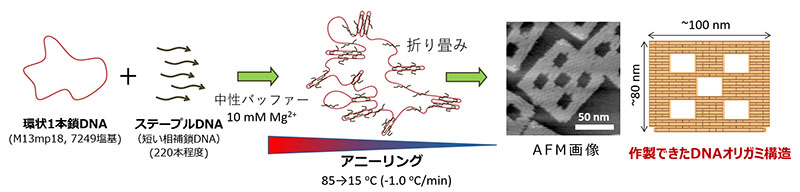
3. 作製したDNAオリガミ

実験手法
原子間力顕微鏡(AFM)走査プローブ顕微鏡の1 つで、試料と鋭い探針の間に働く原子間力を利用して,試料表面の形状を測定する。探針はカンチレバー(板状のバネ)の先端に取り付けてあり,試料との間に原子間力が働くとカンチレバーがたわむので,その変位をカンチレバー背面へのレーザー照射とその反射光の検出によって測定する。試料台に取り付けられた圧電素子の制御によって試料をナノメートルスケールで走査できる。また、カンチレバーを小型化し共鳴周波数を上げることで高速スキャンが可能である。
光ピンセット (Optical Tweezers)
光を回折限界まで収束させることで、水溶液中でその集光点に微粒子を捕捉する方法で、その集光点を移動させることで微粒子を3次元に操作することができる。通常レーザーを光源に用い、集光点に微粒子を捕捉するため、レーザートラッピングとも呼ばれる。開発者のArthur Ashkinは2018年ノーベル物理学賞を受賞した。
ナノポアシーケンシング (Nanopore Sequencing)
DNA,RNA,ペプチドなどの生体高分子の配列を読み出すための新たな技術である。脂質膜に貫通させた数ナノメートルの細孔(ナノポア)を使い、生体高分子がナノポアを通過する際の電流値の変化や通過時間を計測して、これらのデータの機械学習によって塩基配列を決定する。ハイスループットで装置の小型化が可能である。
History of Structural DNA Nanotechnology

James Watson and Francis Crick with Double-helical DNA model
University of Cambridge, May 1953

James Watson, The Discoverer of DNA Structure
Lecture at Harvard University, September 1999
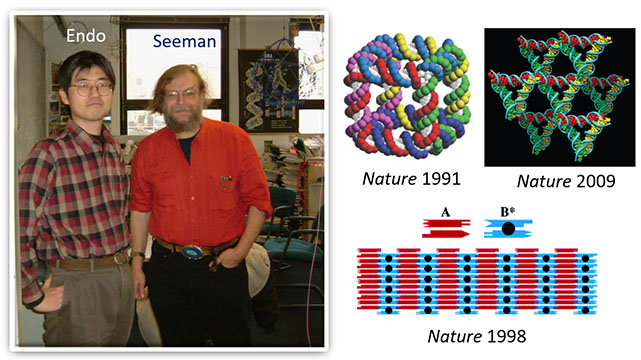
Ned Seeman, The Pioneer of DNA Nanotechnology
New York University, February 2005

Paul Rothemund, The Creator of DNA Origami
Snowbird, UT, USA, April 2010
バナースペース
DNAナノテクノロジーグループ
京都大学高等研究院
物質-細胞統合システム拠点
京都大学吉田西キャンパス
高等研究院本館
〒606-8501
京都市左京区吉田牛ノ宮町
